Phản ứng tổng hợp chuỗi polymerase thời gian thực
Phản ứng chuỗi polymerase thời gian thực (hay PCR thời gian thực, hoặc khi sử dụng để định lượng có thể gọi là qPCR) là một kỹ thuật thí nghiệm trong lĩnh vực sinh học phân tử dựa trên dựa trên phản ứng chuỗi polymerase (PCR). Phương pháp này giám sát quá trình khuếch đại một đoạn DNA cụ thể trong suốt quá trình PCR (tức là thời gian thực), không phải ở cuối quá trình như trong PCR thông thường. PCR thời gian thực có thể được sử dụng định lượng (PCR thời gian thực định lượng) và bán định lượng (nghĩa là trên/dưới một lượng phân tử DNA nhất định) (PCR thời gian thực bán định lượng).
Hai phương pháp phổ biến để phát hiện các sản phẩm PCR trong PCR thời gian thực là (1) thuốc nhuộm huỳnh quang không đặc hiệu xen kẽ với bất kỳ DNA sợi kép và (2) đầu dò DNA đặc hiệu gồm oligonucleotide được dán nhãn bằng máy phóng huỳnh quang, cho phép phát hiện chỉ sau khi lai acid nucleic của đầu dò với trình tự bổ sung của nó.
Hướng dẫn "The Minimum Information for Publication of Quantitative Real-Time PCR Experiments" (MIQE) đề xuất rằng viết tắt "qPCR" được sử dụng để chỉ "quantitative real-time PCR," và "RT-qPCR" được sử dụng để chỉ "reverse transcription–qPCR."[1] Thuật ngữ "RT-PCR" thường được sử dụng để chỉ "phản ứng tổng hợp chuỗi polymerase sao chép ngược" và không phải là "real-time PCR." Tuy nhiên, không tất cả các tác giả đều tuân thủ quy ước này.[2]
Nền tảng
[sửa | sửa mã nguồn]
Tế bào ở tất cả các sinh vật điều hòa biểu hiện gen bằng cách luân chuyển các bản phiên mã gen (chuỗi đơn RNA): Số lượng gen biểu hiện trong tế bào có thể được đo bằng số lượng bản sao của bản phiên mã RNA của gen đó có mặt trong một mẫu. Để phát hiện và định lượng mạnh mẽ biểu hiện gen từ một lượng nhỏ RNA, việc khuếch đại bản phiên mã gen là cần thiết. Phản ứng chuỗi polymerase (PCR) là phương pháp phổ biến để khuếch đại DNA; đối với PCR dựa trên RNA, mẫu RNA trước tiên được sao chép ngược thành DNA bổ sung (cDNA) với Enzyme phiên mã ngược.
Để khuếch đại một lượng nhỏ DNA, phương pháp tương tự được sử dụng như trong PCR thông thường sử dụng mẫu DNA, ít nhất một cặp mồi cụ thể, deoxyribonucleotide triphosphate, một loại thích hợp dung dịch đệm và DNA polymerase ổn định nhiệt. Một chất được đánh dấu bằng fluorophore được thêm vào hỗn hợp này trong máy quay vòng nhiệt có chứa cảm biến để đo huỳnh quang của fluorophore sau khi nó được kích thích ở mức yêu cầu. Bước sóng cho phép đo tốc độ tạo ra cho một hoặc nhiều sản phẩm cụ thể. Điều này cho phép đo tốc độ tạo ra sản phẩm khuếch đại ở mỗi chu kỳ PCR. Do đó, dữ liệu được tạo ra có thể được phân tích bằng phần mềm máy tính để tính toán biểu hiện gen tương đối (hoặc số bản sao mRNA) trong một số mẫu. PCR định lượng cũng có thể được áp dụng để phát hiện và định lượng DNA trong các mẫu để xác định sự hiện diện và độ phong phú của một chuỗi DNA cụ thể trong các mẫu này.[3] Phép đo này được thực hiện sau mỗi chu kỳ khuếch đại và đây là lý do tại sao phương pháp này được gọi là PCR thời gian thực (nghĩa là PCR ngay lập tức hoặc đồng thời).
PCR định lượng và DNA microarray là các phương pháp hiện đại để nghiên cứu biểu hiện gen. Các phương pháp cũ hơn đã được sử dụng để đo mức độ phong phú của mRNA: hiển thị vi sai, xét nghiệm bảo vệ RNase và phương pháp thấm northern. Phương pháp thấm northern thường được sử dụng để ước tính mức độ biểu hiện của gen bằng cách hình dung mức độ phong phú của bản phiên mã mRNA của nó trong một mẫu. Trong phương pháp này, RNA tinh khiết được phân tách bằng điện di trên gel agarose, chuyển sang nền rắn (chẳng hạn như màng nylon) và được thăm dò bằng đầu dò DNA hoặc RNA cụ thể là bổ sung cho gen quan tâm. Mặc dù kỹ thuật này vẫn được sử dụng để đánh giá biểu hiện gen nhưng nó đòi hỏi lượng RNA tương đối lớn và chỉ cung cấp thông tin định tính hoặc bán định lượng về mức độ mRNA.[4] Lỗi ước tính phát sinh từ những thay đổi trong phương pháp định lượng có thể là kết quả của tính toàn vẹn DNA, hiệu suất enzyme và nhiều yếu tố khác. Vì lý do này, một số hệ thống tiêu chuẩn hóa (thường được gọi là phương pháp chuẩn hóa) đã được phát triển. Một số đã được phát triển để định lượng tổng số biểu hiện gen, nhưng phổ biến nhất là nhằm mục đích định lượng gen cụ thể đang được nghiên cứu liên quan đến một gen khác gọi là gen bình thường hóa, được chọn lọc để có mức độ biểu hiện gần như không đổi. Những gen này thường được chọn từ gen quản lý vì chức năng của chúng liên quan đến sự sống sót cơ bản của tế bào thường bao hàm biểu hiện gen cấu thành.[5][6] Điều này cho phép các nhà nghiên cứu báo cáo tỷ lệ biểu hiện của các gen quan tâm chia cho biểu hiện của bộ chuẩn hóa đã chọn, từ đó cho phép so sánh bộ chuẩn hóa trước đó mà không thực sự biết mức độ biểu hiện tuyệt đối của nó.
Các gen chuẩn hóa được sử dụng phổ biến nhất là những gen mã hóa cho các phân tử sau: tubulin, glyceraldehyde-3-phosphate dehydrogenase, albumin, cyclophilin và các RNA ribosome.[4]
Các nguyên tắc cơ bản
[sửa | sửa mã nguồn]PCR thời gian thực được thực hiện trong máy quay vòng nhiệt có khả năng chiếu sáng từng mẫu bằng một chùm ánh sáng có ít nhất một bước sóng xác định và phát hiện huỳnh quang phát ra từ fluorophore bị kích thích. Máy chu trình nhiệt cũng có thể làm nóng và làm lạnh mẫu nhanh chóng, nhờ đó tận dụng được các đặc tính hóa lý của acid nucleic và DNA polymerase.
Quá trình PCR thường bao gồm một loạt các thay đổi nhiệt độ được lặp lại 25–50 lần. Các chu trình này thường bao gồm ba giai đoạn: giai đoạn thứ nhất, ở khoảng 95 °C, cho phép tách chuỗi kép acid nucleic; thứ hai, ở nhiệt độ khoảng 50–60 °C, cho phép liên kết mồi với mẫu DNA;[7] thứ ba, ở nhiệt độ từ 68 đến 72 °C, tạo điều kiện thuận lợi cho phản ứng trùng hợp được thực hiện bởi DNA polymerase. Do kích thước nhỏ của các đoạn, bước cuối cùng thường bị bỏ qua trong loại PCR này vì enzyme có thể sao chép DNA amplicon trong quá trình thay đổi giữa giai đoạn căn chỉnh và giai đoạn biến tính. Ngoài ra, trong PCR bốn bước, huỳnh quang được đo trong các giai đoạn nhiệt độ ngắn chỉ kéo dài vài giây trong mỗi chu kỳ, với nhiệt độ, ví dụ: 80 °C, để giảm tín hiệu gây ra bởi sự hiện diện của chất điều chỉnh mồi khi sử dụng thuốc nhuộm không đặc hiệu.[8] Nhiệt độ và thời gian được sử dụng cho mỗi chu kỳ phụ thuộc vào nhiều thông số khác nhau, chẳng hạn như: enzyme được sử dụng để tổng hợp DNA, nồng độ của các ion hóa trị hai và deoxyribonucleotide triphosphate (dNTPs) trong phản ứng và nhiệt độ liên kết của các mồi.[9]
Phân loại hóa học
[sửa | sửa mã nguồn]Kỹ thuật PCR thời gian thực có thể được phân loại theo hóa chất được sử dụng để phát hiện sản phẩm PCR, fluorochromes cụ thể hoặc không đặc hiệu.
Phát hiện không đặc hiệu: PCR thời gian thực với thuốc nhuộm liên kết DNA sợi đôi làm chất thăm dò
[sửa | sửa mã nguồn]Thuốc nhuộm liên kết DNA liên kết với tất cả (ds) DNA sợi đôi trong PCR, làm tăng hiệu suất lượng tử huỳnh quang của thuốc nhuộm. Do đó, sự gia tăng sản phẩm DNA trong quá trình PCR dẫn đến tăng cường độ huỳnh quang đo được ở mỗi chu kỳ. Tuy nhiên, thuốc nhuộm DSDNA như SYBR Green sẽ liên kết với tất cả các sản phẩm PCR DSDNA, bao gồm cả các sản phẩm PCR không đặc hiệu (chẳng hạn như primer dimer). Điều này có khả năng gây trở ngại hoặc ngăn chặn việc giám sát chính xác trình tự mục tiêu đã định.
Trong PCR thời gian thực với thuốc nhuộm DSDNA, phản ứng được chuẩn bị như bình thường với việc bổ sung thuốc nhuộm DSDNA huỳnh quang. Sau đó, phản ứng được thực hiện trong công cụ PCR thời gian thực và sau mỗi chu kỳ, cường độ huỳnh quang được đo bằng máy dò; thuốc nhuộm chỉ phát huỳnh quang khi liên kết với DSDNA (tức là sản phẩm PCR). Phương pháp này có ưu điểm là chỉ cần một cặp mồi để thực hiện khuếch đại, giúp giảm chi phí; nhiều chuỗi mục tiêu có thể được theo dõi trong một ống bằng cách sử dụng các loại thuốc nhuộm khác nhau.
Phát hiện cụ thể: phương pháp đầu dò chỉ thị huỳnh quang
[sửa | sửa mã nguồn]
Đầu dò chỉ thị huỳnh quang chỉ phát hiện DNA chứa trình tự bổ sung cho đầu dò; do đó, việc sử dụng đầu dò chỉ thị làm tăng đáng kể độ đặc hiệu và cho phép thực hiện kỹ thuật này ngay cả khi có sự hiện diện của DSDNA khác. Sử dụng các nhãn có màu khác nhau, đầu dò chỉ thị huỳnh quang có thể được sử dụng trong các xét nghiệm ghép kênh để theo dõi một số chuỗi mục tiêu trong cùng một ống. Độ đặc hiệu của đầu dò chỉ thị huỳnh quang cũng ngăn chặn sự can thiệp của các phép đo gây ra bởi các primer dimer, là những sản phẩm phụ tiềm ẩn không mong muốn trong PCR. Tuy nhiên, đầu dò chỉ thị huỳnh quang không ngăn được tác dụng ức chế của các primer dimer, điều này có thể làm giảm sự tích tụ của các sản phẩm mong muốn trong phản ứng.
Phương pháp này dựa trên đầu dò DNA với chất chỉ thị huỳnh quang ở một đầu và dập tắt huỳnh quang ở đầu đối diện của đầu dò. Sự ở gần của đầu dò với chất khử chất khử sẽ ngăn cản việc phát hiện huỳnh quang của nó; sự phá vỡ đầu dò theo hoạt động 5' đến 3' exonuclease của Taq polymerase phá vỡ khoảng cách gần chất chỉ thị-chất dập tắt và do đó cho phép phát huỳnh quang không bị dập tắt, có thể được phát hiện sau kích thích bằng tia laze. Do đó, sự gia tăng sản phẩm được nhắm mục tiêu bởi đầu dò chỉ thị ở mỗi chu kỳ PCR gây ra sự gia tăng huỳnh quang theo tỷ lệ do sự cố của đầu dò và giải phóng đầu báo cáo.
- PCR được chuẩn bị như bình thường (xem PCR), và đầu dò báo cáo được thêm vào.
- Khi phản ứng bắt đầu, trong giai đoạn ủ của PCR cả đầu dò và mồi đều gắn vào DNA đích.
- Quá trình trùng hợp của chuỗi DNA mới được bắt đầu từ các đoạn mồi và khi polymerase tiếp cận đầu dò, 5'-3'-exonuclease của nó sẽ làm suy giảm đầu dò, tách vật lý chất phát huỳnh quang khỏi chất khử chất khử, dẫn đến tăng huỳnh quang.
- Huỳnh quang được phát hiện và đo bằng máy PCR thời gian thực và mức tăng hình học của nó tương ứng với mức tăng theo cấp số nhân của sản phẩm được sử dụng để xác định chu kỳ định lượng (Cq ) trong mỗi phản ứng.
Phân tích nhiệt độ dung hợp
[sửa | sửa mã nguồn]
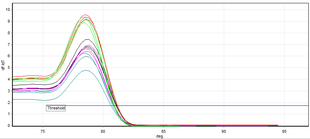
PCR thời gian thực cho phép xác định các đoạn DNA cụ thể, được khuếch đại bằng cách sử dụng phân tích nhiệt độ nóng chảy của chúng (còn gọi là giá trị Tm, từ melting nhiệt độ). Phương pháp được sử dụng thường là PCR với thuốc nhuộm liên kết DNA sợi đôi làm phóng viên và thuốc nhuộm được sử dụng thường là SYBR Green. Nhiệt độ nóng chảy DNA đặc trưng cho đoạn được khuếch đại. Kết quả của kỹ thuật này thu được bằng cách so sánh các đường cong phân ly của các mẫu DNA được phân tích.[11]
Không giống như PCR thông thường, phương pháp này tránh sử dụng kỹ thuật điện di trước đây để chứng minh kết quả của tất cả các mẫu. Điều này là do, mặc dù là một kỹ thuật động học, PCR định lượng thường được đánh giá ở một điểm cuối khác biệt. Do đó, kỹ thuật này thường cho kết quả nhanh hơn và/hoặc sử dụng ít chất phản ứng hơn so với điện di. Nếu cần phải điện di tiếp theo thì chỉ cần kiểm tra những mẫu mà PCR thời gian thực cho thấy có nghi ngờ và/hoặc phê chuẩn kết quả đối với các mẫu đã xét nghiệm dương tính với một yếu tố xác định cụ thể.
Mô hình hóa
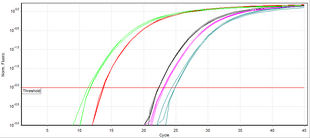
[sửa | sửa mã nguồn]Không giống như PCR điểm cuối (PCR thông thường), PCR thời gian thực cho phép theo dõi sản phẩm mong muốn tại bất kỳ thời điểm nào trong quá trình khuếch đại bằng cách đo huỳnh quang (trong khung thời gian thực, phép đo được thực hiện ở mức của nó trên một ngưỡng nhất định). Một phương pháp định lượng DNA thường được sử dụng bằng PCR thời gian thực dựa vào việc lập biểu đồ huỳnh quang theo số chu kỳ trên thang logarit. Ngưỡng phát hiện huỳnh quang dựa trên DNA được đặt 3–5 lần độ lệch chuẩn của nhiễu tín hiệu trên nền. Số chu kỳ mà huỳnh quang vượt quá ngưỡng được gọi là chu kỳ ngưỡng (Ct) hoặc theo hướng dẫn MIQE, chu kỳ định lượng (C q).[1]
Trong giai đoạn khuếch đại theo cấp số nhân, số lượng mẫu DNA đích (khuếch đại) tăng gấp đôi sau mỗi chu kỳ. Ví dụ: một mẫu DNA có Cq đi trước mẫu khác 3 chu kỳ chứa mẫu nhiều hơn 23 = 8 lần. Tuy nhiên, hiệu quả khuếch đại thường khác nhau giữa các đoạn mồi và mẫu. Do đó, hiệu quả của sự kết hợp mồi-mẫu được đánh giá trong thí nghiệm chuẩn độ với các pha loãng nối tiếp của mẫu DNA để tạo ra đường cong chuẩn về sự thay đổi trong (C< sub>q) với mỗi độ pha loãng. Sau đó, độ dốc của hồi quy tuyến tính được sử dụng để xác định hiệu suất khuếch đại, là 100% nếu độ pha loãng 1:2 dẫn đến (Cq) chênh lệch bằng 1. Phương pháp ngưỡng chu kỳ đưa ra một số giả định về cơ chế phản ứng và dựa vào dữ liệu từ các vùng tín hiệu trên tạp âm thấp của cấu hình khuếch đại có thể tạo ra sự khác biệt đáng kể trong quá trình phân tích dữ liệu .[12]
Để định lượng biểu hiện gen, (Cq) đối với RNA hoặc DNA từ gen quan tâm sẽ được trừ khỏi ( Cq) của RNA/DNA từ gen giữ nhà trong cùng một mẫu để chuẩn hóa sự thay đổi về số lượng và chất lượng RNA giữa các mẫu khác nhau. Quy trình chuẩn hóa này thường được gọi là phương thức ΔCt[13] và cho phép so sánh sự biểu hiện của gen quan tâm giữa các mẫu khác nhau. Tuy nhiên, để so sánh như vậy, sự biểu hiện của gen tham chiếu chuẩn hóa cần phải rất giống nhau trên tất cả các mẫu. Do đó, việc chọn một gen tham chiếu đáp ứng tiêu chí này có tầm quan trọng cao và thường là thách thức vì chỉ có rất ít gen thể hiện mức độ biểu hiện như nhau trên nhiều tình trạng hoặc mô khác nhau.[14][15] Mặc dù phân tích ngưỡng chu kỳ được tích hợp với nhiều hệ thống phần mềm thương mại, nhưng có các phương pháp phân tích dữ liệu hồ sơ khuếch đại chính xác và đáng tin cậy hơn cần được xem xét trong trường hợp cần quan tâm đến khả năng tái tạo.[12]
Các phương pháp định lượng qPCR dựa trên cơ chế cũng đã được đề xuất và có ưu điểm là chúng không yêu cầu đường cong chuẩn để định lượng. Các phương pháp như MAK2[16] đã được chứng minh là có hiệu suất định lượng tương đương hoặc tốt hơn so với các phương pháp đường cong tiêu chuẩn. Các phương pháp dựa trên cơ chế này sử dụng kiến thức về quá trình khuếch đại polymerase để ước tính nồng độ mẫu ban đầu. Phần mở rộng của phương pháp này bao gồm một mô hình chính xác của toàn bộ cấu hình phản ứng PCR, cho phép sử dụng dữ liệu có tỷ lệ tín hiệu trên nhiễu cao và khả năng xác thực chất lượng dữ liệu trước khi phân tích.[12]
Theo nghiên cứu của Ruijter et al.[17] MAK2 giả định hiệu suất khuếch đại không đổi trong quá trình phản ứng PCR. Tuy nhiên, phân tích lý thuyết về phản ứng chuỗi polymerase, từ đó tạo ra MAK2, đã tiết lộ rằng hiệu suất khuếch đại không cố định trong suốt PCR. Trong khi định lượng MAK2 cung cấp ước tính đáng tin cậy về nồng độ DNA mục tiêu trong mẫu trong điều kiện qPCR bình thường, thì MAK2 không định lượng đáng tin cậy nồng độ mục tiêu cho xét nghiệm qPCR bằng máy đo cạnh tranh.
Các ứng dụng
[sửa | sửa mã nguồn]Có rất nhiều ứng dụng cho phản ứng chuỗi polymerase định lượng trong phòng thí nghiệm. Nó thường được sử dụng cho cả chẩn đoán và nghiên cứu cơ bản. Ứng dụng của kỹ thuật này trong công nghiệp bao gồm định lượng lượng vi sinh vật trong thực phẩm hoặc trên thực vật, phát hiện GMO (sinh vật biến đổi gen) cũng như định lượng và xác định kiểu gen của mầm bệnh virus ở người.
Định lượng biểu hiện gen
[sửa | sửa mã nguồn]Định lượng biểu hiện gen bằng các phương pháp phát hiện DNA truyền thống là không đáng tin cậy. Việc phát hiện mRNA trên northern blot hoặc sản phẩm PCR trên gel hoặc Southern blot không cho phép định lượng chính xác.[18] Ví dụ: trong 20–40 chu kỳ của một PCR thông thường, lượng sản phẩm DNA đạt đến bình nguyên không tương quan trực tiếp với lượng DNA mục tiêu trong PCR ban đầu.[19]
PCR thời gian thực có thể được sử dụng để định lượng acid nucleic bằng hai phương pháp phổ biến: định lượng tương đối và định lượng tuyệt đối.[20] Định lượng tuyệt đối đưa ra số lượng phân tử DNA mục tiêu chính xác bằng cách so sánh với các tiêu chuẩn DNA bằng cách sử dụng đường cong hiệu chuẩn. Do đó, điều cần thiết là PCR của mẫu và chất chuẩn phải có cùng hiệu suất khuếch đại.[21] Định lượng tương đối dựa trên các gen tham chiếu nội bộ để xác định sự khác biệt gấp bội trong biểu hiện của gen mục tiêu. Việc định lượng được biểu thị bằng sự thay đổi mức độ biểu hiện của mRNA được hiểu là DNA bổ sung (cDNA, được tạo ra bởi reverse Transcription của mRNA). Việc định lượng tương đối dễ thực hiện hơn vì nó không yêu cầu đường chuẩn vì lượng gen được nghiên cứu được so sánh với lượng gen tham chiếu đối chứng.
Vì đơn vị được sử dụng để biểu thị kết quả định lượng tương đối không quan trọng nên kết quả có thể được so sánh trên một số RTqPCR khác nhau. Lý do sử dụng một hoặc nhiều gen giữ nhà là để điều chỉnh các biến thể không đặc hiệu, chẳng hạn như sự khác biệt về số lượng và chất lượng RNA được sử dụng, điều này có thể ảnh hưởng đến hiệu quả của quá trình sao chép ngược và do đó ảnh hưởng đến hiệu quả của toàn bộ quá trình PCR. Tuy nhiên, khía cạnh quan trọng nhất của quá trình này là gen tham chiếu phải ổn định.[22]
Việc lựa chọn các gen tham chiếu này theo truyền thống được thực hiện trong sinh học phân tử bằng cách sử dụng các nghiên cứu định tính hoặc bán định lượng như kiểm tra trực quan gel RNA, Northern blot đo mật độ hoặc PCR bán định lượng (mô phỏng PCR). Giờ đây, trong kỷ nguyên gen, có thể thực hiện ước tính chi tiết hơn cho nhiều sinh vật bằng cách sử dụng công nghệ phiên mã.[23] Tuy nhiên, nghiên cứu đã chỉ ra rằng mức khuếch đại của phần lớn các gen tham chiếu được sử dụng để định lượng biểu hiện của mRNA thay đổi tùy theo điều kiện thí nghiệm.[24][25][26] Do đó, cần phải thực hiện nghiên cứu phương pháp luận hợp lý thống kê ban đầu để chọn ra gen tham chiếu phù hợp nhất.
Một số thuật toán thống kê đã được phát triển có thể phát hiện gen nào hoặc các gen nào phù hợp nhất để sử dụng trong những điều kiện nhất định. Những thứ như geNORM hoặc BestKeeper có thể so sánh các cặp hoặc trung bình hình học cho một ma trận gồm các gen tham chiếu khác nhau và mô.[4][6]
Công dụng chẩn đoán
[sửa | sửa mã nguồn]PCR định tính chẩn đoán được áp dụng để phát hiện nhanh chóng acid nucleic được chẩn đoán, ví dụ: bệnh truyền nhiễm,[27][28] ung thư và các bất thường về di truyền. Việc đưa các xét nghiệm PCR định tính vào phòng thí nghiệm vi sinh lâm sàng đã cải thiện đáng kể việc chẩn đoán các bệnh truyền nhiễm,[29] và được triển khai như một công cụ để phát hiện các bệnh mới nổi, chẳng hạn như các chủng flu và coronavirus mới,[30] trong xét nghiệm chẩn đoán.[31][32]
Công dụng vi sinh
[sửa | sửa mã nguồn]PCR định lượng cũng được sử dụng bởi các nhà vi trùng học làm việc trong lĩnh vực an toàn thực phẩm, hư hỏng và lên men thực phẩm cũng như đánh giá rủi ro vi khuẩn đối với chất lượng nước (nước uống và nước giải khát) cũng như bảo vệ sức khỏe cộng đồng.[33]
qPCR cũng có thể được sử dụng để khuếch đại các dấu hiệu phân loại hoặc chức năng của gen trong DNA lấy từ các mẫu môi trường.[34] Các điểm đánh dấu được thể hiện bằng các đoạn DNA di truyền hoặc DNA bổ sung.[34] Bằng cách khuếch đại một yếu tố di truyền nhất định, người ta có thể định lượng lượng nguyên tố đó trong mẫu trước khi khuếch đại.[34] Sử dụng các dấu hiệu phân loại (gen ribosome) và qPCR có thể giúp xác định số lượng vi sinh vật trong một mẫu và có thể xác định các họ, chi hoặc loài khác nhau dựa trên tính đặc hiệu của dấu hiệu.[34] Sử dụng các dấu hiệu chức năng (gen mã hóa protein) có thể hiển thị biểu hiện gen trong cộng đồng, điều này có thể tiết lộ thông tin về môi trường.[34]
Phát hiện mầm bệnh thực vật
[sửa | sửa mã nguồn]Ngành nông nghiệp không ngừng nỗ lực tạo ra cây giống hoặc cây giống không có mầm bệnh nhằm ngăn ngừa thiệt hại kinh tế và bảo vệ sức khỏe. Các hệ thống đã được phát triển cho phép phát hiện một lượng nhỏ DNA của Phytophthora ramorum, một loại nấm oomycete có thể giết chết oaks và các loài khác, trộn lẫn với DNA của cây chủ. Việc phân biệt DNA của mầm bệnh và thực vật dựa trên sự khuếch đại các trình tự ITS, các đoạn đệm nằm trong vùng mã hóa của gen ribosomal RNA, đặc trưng cho từng taxon.[35] Các phiên bản thực địa của kỹ thuật này cũng đã được phát triển để xác định mầm bệnh tương tự.[36]
Phát hiện sinh vật biến đổi gen
[sửa | sửa mã nguồn]qPCR sử dụng phiên mã ngược (RT-qPCR) có thể được sử dụng để phát hiện GMO nhờ độ nhạy và phạm vi động trong việc phát hiện DNA. Các phương pháp thay thế như phân tích DNA hoặc protein thường ít nhạy cảm hơn. Các đoạn mồi cụ thể được sử dụng để khuếch đại không phải gen chuyển mà là promoter, terminator hoặc thậm chí các trình tự trung gian được sử dụng trong quá trình thiết kế vectơ. Vì quá trình tạo ra cây chuyển gen thường dẫn đến việc chèn nhiều hơn một bản sao của gen chuyển nên số lượng của nó cũng thường được đánh giá. Điều này thường được thực hiện bằng cách định lượng tương đối bằng cách sử dụng một gen đối chứng từ các loài được xử lý chỉ hiện diện dưới dạng một bản sao duy nhất.[37][38]
Định lượng lâm sàng và kiểu gen
[sửa | sửa mã nguồn]Vi-rút có thể hiện diện ở người do nhiễm trùng trực tiếp hoặc đồng nhiễm, khiến chẩn đoán khó sử dụng các kỹ thuật cổ điển và có thể dẫn đến tiên lượng và điều trị không chính xác. Việc sử dụng qPCR cho phép cả định lượng và xác định kiểu gen (đặc tính của chủng, được thực hiện bằng cách sử dụng các đường cong nóng chảy) của một loại vi-rút như vi-rút viêm gan B.[39] Mức độ lây nhiễm, được định lượng bằng các bản sao của bộ gen virus trên một đơn vị mô của bệnh nhân, có liên quan trong nhiều trường hợp; ví dụ: xác suất herpes simplex virus loại 1 kích hoạt lại có liên quan đến số lượng tế bào thần kinh bị nhiễm trong hạch.[40] Việc định lượng này được thực hiện bằng phiên mã ngược hoặc không có phiên mã ngược, như xảy ra nếu vi rút tích hợp vào bộ gen của con người tại bất kỳ thời điểm nào trong chu kỳ của nó, chẳng hạn như xảy ra trong trường hợp HPV (vi rút u nhú ở người), trong đó một số các biến thể của nó có liên quan đến sự xuất hiện của ung thư cổ tử cung.[41] PCR thời gian thực cũng đã mang lại sự định lượng human cytomegalovirus (CMV) được thấy ở những bệnh nhân bị ức chế miễn dịch sau ghép tạng hoặc tủy xương.[42]
Tham khảo
[sửa | sửa mã nguồn]- ^ a b Bustin SA, Benes V, Garson JA, Hellemans J, Huggett J, Kubista M, Mueller R, Nolan T, Pfaffl MW, Shipley GL, Vandesompele J, Wittwer CT (2009). “The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments”. Clinical Chemistry. 55 (4): 611–622. doi:10.1373/clinchem.2008.112797. PMID 19246619.
- ^ Logan, Julie; Edwards, Kirstin & Saunders, Nick biên tập (2009). Real-Time PCR: Current Technology and Applications. Caister Academic Press. ISBN 978-1-904455-39-4.
- ^ Watson, J D; Baker, T A; Bell, S P; Gann, A; Levine, M; Losick, R (2004). Molecular Biology of the Gene . San Francisco: Benjamin Cummings. ISBN 978-0-321-22368-5.
- ^ a b c Pfaffl, MW; Tichopad, A; Prgomet, C; Neuvians, TP (tháng 3 năm 2004). “Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper--Excel-based tool using pair-wise correlations”. Biotechnology Letters. 26 (6): 509–15. doi:10.1023/b:bile.0000019559.84305.47. PMID 15127793. S2CID 977404.
- ^ Pfaffl, MW; Horgan, GW; Dempfle, L (2002). “Relative Expression Software Tool (REST©) for group wise comparison and statistical analysis of relative expression results in real-time PCR”. Nucleic Acids Res. 30 (9): e36. doi:10.1093/nar/30.9.e36. PMC 113859. PMID 11972351.
- ^ a b Vandesompele, J; De Preter, K; Pattyn, F; Poppe, B; Van Roy, N; De Paepe, A; Speleman, F (2002). “Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes”. Genome Biology. 3 (7): 1–12. doi:10.1186/gb-2002-3-7-research0034. PMC 126239. PMID 12184808.
- ^ Rychlik W, Spencer WJ, Rhoads RE (1990). “Optimization of the annealing temperature for DNA amplification in vitro”. Nucleic Acids Res. 18 (21): 6409–6412. doi:10.1093/nar/18.21.6409. PMC 332522. PMID 2243783.
- ^ Pfaffl, Michael (2000). “Development and Validation of an Externally Standardized Quantitative Insulin-like Growth Factor-1 RT-PCR Using LightCycler SYBR Green I Technology” (PDF). Biochemica (3) – qua gene-quantification.org.
- ^ Sambrook, Joseph; Russel, David W. (2001). Molecular Cloning: A Laboratory Manual (ấn bản thứ 3). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-576-7.
- ^ Ponchel F; Toomes C; Bransfield K; Leong F.T; Douglas S.H; Field S.L; Bell S.M; Combaret V; Puisieux A; Mighell A.J (2003). “Real-time PCR based on SYBR-Green I fluorescence: An alternative to the TaqMan assay for a relative quantification of gene rearrangements, gene amplifications and micro gene deletions”. BMC Biotechnol. 3: 18. doi:10.1186/1472-6750-3-18. PMC 270040. PMID 14552656.
- ^ Ririe K.M; Rasmussen R.P; Wittwer C.T. (1997). “Product Differentiation by Analysis of DNA Melting Curves during the Polymerase Chain Reaction” (PDF). Analytical Biochemistry. 245 (2): 154–160. doi:10.1006/abio.1996.9916. PMID 9056205. Bản gốc (PDF) lưu trữ ngày 5 tháng 8 năm 2004. Truy cập ngày 4 tháng 12 năm 2023.
- ^ a b c Carr, A. C.; Moore, S. D. (2012). Lucia, Alejandro (biên tập). “Robust Quantification of Polymerase Chain Reactions Using Global Fitting”. PLOS ONE. 7 (5): e37640. Bibcode:2012PLoSO...737640C. doi:10.1371/journal.pone.0037640. PMC 3365123. PMID 22701526.
- ^ Schefe JH, Lehmann KE, Buschmann IR, Unger T, Funke-Kaiser H (2006). “Quantitative real-time RT-PCR data analysis: current concepts and the novel "gene expression's CT difference" formula”. J Mol Med. 84 (11): 901–910. doi:10.1007/s00109-006-0097-6. PMID 16972087. S2CID 10427299.
- ^ Nailis H, Coenye T, Van Nieuwerburgh F, Deforce D, Nelis HJ (2006). “Development and evaluation of different normalization strategies for gene expression studies in Candida albicans biofilms by real-time PCR”. BMC Mol. Biol. 7 (1): 25. doi:10.1186/1471-2199-7-25. PMC 1557526. PMID 16889665.
- ^ Nolan T, Hands RE, Bustin SA (2006). “Quantification of mRNA using real-time RT-PCR”. Nat. Protoc. 1 (3): 1559–1582. doi:10.1038/nprot.2006.236. PMID 17406449. S2CID 10108148.
- ^ Boggy G, Woolf PJ (2010). Ravasi T (biên tập). “A Mechanistic Model of PCR for Accurate Quantification of Quantitative PCR Data”. PLOS ONE. 5 (8): e12355. Bibcode:2010PLoSO...512355B. doi:10.1371/journal.pone.0012355. PMC 2930010. PMID 20814578.
- ^ Ruijter JM, Pfaffl MW, Zhao S, Spiess AN, Boggy G, Blom J, Rutledge RG, Sisti D, Lievens A, De Preter K, Derveaux S, Hellemans J, Vandesompele J (2012). “Evaluation of qPCR curve analysis methods for reliable biomarker discovery: bias, resolution, precision, and implications”. Methods. 59 (1): 32–46. doi:10.1016/j.ymeth.2012.08.011. PMID 22975077.
- ^ Bruce Gelerter. “PEMF For Treatment Of Corneal Disorders”. lemuriatechnologies.com. Bản gốc lưu trữ ngày 9 tháng 6 năm 2014.
- ^ Overbergh, L.; Giulietti, A.; Valckx, D.; Decallonne, R.; Bouillon, R.; Mathieu, C. (2003). “The use of real-time reverse transcriptase PCR for the quantification of cytokine gene expression”. Journal of Biomolecular Techniques. 14 (1): 33–43. PMC 2279895. PMID 12901609.
- ^ S. Dhanasekaran; T. Mark Doherty; John Kenneth; TB Trials Study Group (tháng 3 năm 2010). “Comparison of different standards for real-time PCR-based absolute quantification”. Journal of Immunological Methods. 354 (1–2): 34–39. doi:10.1016/j.jim.2010.01.004. PMID 20109462.
- ^ Bar, Tzachi; Kubista, Mikael; Tichopad, Ales (19 tháng 10 năm 2011). “Validation of kinetics similarity in qPCR”. Nucleic Acids Research (bằng tiếng Anh). 40 (4): 1395–1406. doi:10.1093/nar/gkr778. ISSN 0305-1048. PMC 3287174. PMID 22013160.
- ^ Brunner, AM; Yakovlev, IA; Strauss, SH (2004). “Validating internal controls for quantitative plant gene expression studies”. BMC Plant Biol. 4: 14. doi:10.1186/1471-2229-4-14. PMC 515301. PMID 15317655.
- ^ McGettigan, Paul A (2013). “Transcriptomics in the RNA-seq era”. Current Opinion in Chemical Biology. 17 (1): 4–11. doi:10.1016/j.cbpa.2012.12.008. PMID 23290152.
- ^ Thellin, O; Zorzi, W; Lakaye, B; De Borman, B; Coumans, B; Henne, G; Grisar, T; Igout, A; Heinen, E (1999). “Housekeeping genes as internal standards: use and limits”. J Biotechnol. 75 (2–3): 197–200. doi:10.1016/s0168-1656(99)00163-7. hdl:2268/3661. PMID 10617337.
- ^ Radonic, A; Thulke, S; Mackay, IM; Landt, O; Siegert, W; Nitsche, A (2004). “Guideline for reference gene selection for quantitative real-time PCR”. Biochem Biophys Res Commun. 313 (4): 856–862. doi:10.1016/j.bbrc.2003.11.177. PMID 14706621. Bản gốc lưu trữ ngày 2 tháng 8 năm 2013.
- ^ Dheda, K; Huggett, JF; Bustin, SA; Johnson, MA; Rook, G; Zumla, A (2004). “Validation of housekeeping genes for normalizing RNA expression in real-time PCR”. BioTechniques. 37 (1): 112–119. doi:10.2144/04371RR03. PMID 15283208.
- ^ Jawerth, Nicole (27 tháng 3 năm 2020). “How is the COVID-19 Virus Detected using real time reverse transcription–polymerase chain reaction?”. International Atomic Energy Agency (bằng tiếng Anh). Truy cập ngày 16 tháng 2 năm 2023.
- ^ Xiu, Leshan; Binder, Raquel A.; Alarja, Natalie A.; Kochek, Kara; Coleman, Kristen K.; Than, Son T.; Bailey, Emily S.; Bui, Vuong N.; Toh, Teck-Hock; Erdman, Dean D.; Gray, Gregory C. (tháng 7 năm 2020). “A RT-PCR assay for the detection of coronaviruses from four genera”. Journal of Clinical Virology. 128: 104391. doi:10.1016/j.jcv.2020.104391. PMC 7192118. PMID 32403008.
- ^ Espy, M.J. (tháng 1 năm 2006). “Real-Time PCR in Clinical Microbiology: Applications for Routine Laboratory Testing”. Clinical Microbiology Reviews. 19 (3): 165–256. doi:10.1128/CMR.19.1.165-256.2006. PMC 1360278. PMID 16418529.
- ^ Dhamad, AE; Abdal Rhida, MA (2020). “COVID-19: molecular and serological detection methods”. PeerJ. 8: e10180. doi:10.7717/peerj.10180. PMC 7547594. PMID 33083156.
- ^ “FDA-cleared RT-PCR Assays and Other Molecular Assays for Influenza Viruses” (PDF). cdc.gov.
- ^ “rRT-PCR, a method to confirm Wuhan coronavirus case – Artificial Intelligence for Chemistry” (bằng tiếng Anh). 24 tháng 1 năm 2020. Truy cập ngày 26 tháng 1 năm 2020.
- ^ Filion, M biên tập (2012). Quantitative Real-time PCR in Applied Microbiology. Caister Academic Press. ISBN 978-1-908230-01-0.
- ^ a b c d e Bouchez, Blieux, Dequiedt, Domaizon, Dufresne, Ferreira, Godon, Hellal, Joulian, Quaiser, Martin-Laurent, Mauffret, Monier, Peyret, Schmitt-Koplin, Sibourg, D'oiron, Bispo, Deportes, Grand, Cuny, Maron, Ranjard (tháng 9 năm 2016). “Molecular Microbiology Methods For Environmental Diagnosis”. Environmental Chemistry Letters. 14 (4): 423–441. doi:10.1007/s10311-016-0581-3. S2CID 88827291. Truy cập ngày 11 tháng 5 năm 2020.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^ Baldwin, B.G. (1992). “Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: An example from the Compositaogy”. Molecular Phylogenetics and Evolution. 1 (1): 3–16. doi:10.1016/1055-7903(92)90030-K. PMID 1342921.
- ^ Tomlinson, J. A.; Barker, I.; Boonham, N. (2007). “Faster, Simpler, More-Specific Methods for Improved Molecular Detection of Phytophthora ramorum in the Field”. Applied and Environmental Microbiology. 73 (12): 4040–4047. Bibcode:2007ApEnM..73.4040T. doi:10.1128/AEM.00161-07. PMC 1932743. PMID 17449689.
- ^ Holst-Jensen, Arne; Rønning, Sissel B.; Løvseth, Astrid; Berdal, Knut G. (2003). “PCR technology for screening and quantification of genetically modified organisms (GMOs)”. Analytical and Bioanalytical Chemistry. 375 (8): 985–993. doi:10.1007/s00216-003-1767-7. PMID 12733008. S2CID 43681211.
- ^ Brodmann P.D; Ilg E.C; Berthoud H; Herrmann A. (2002). “... -Time Quantitative Polymerase Chain Reaction Methods for Four Genetically Modified Maize Varieties ...”. Journal of AOAC International. 85 (3): 646–653. doi:10.1093/jaoac/85.3.646. PMID 12083257.
- ^ Yeh S.H. Tsai C.Y. Kao J.H. Liu C.J. Kuo T.J. Lin M.W. Huang W.L. Lu S.F. Jih J. Chen D.S. Others (2004). “Quantification and genotyping of hepatitis B virus in a single reaction by real-time PCR and melting ...”. Journal of Hepatology. 41 (4): 659–666. doi:10.1016/j.jhep.2004.06.031. PMID 15464248.
- ^ Sawtell N.M. (1998). “The Probability of in Vivo Reactivation of Herpes Simplex Virus Type 1 Increases with the Number of Latently Infected Neurons in the Ganglia”. Journal of Virology. 72 (8): 6888–6892. doi:10.1128/JVI.72.8.6888-6892.1998. PMC 109900. PMID 9658140.
- ^ Peter M. Rosty C. Couturier J. Radvanyi F. Teshima H. Sastre-garau X. (2006). “MYC activation associated with the integration of HPV DNA at the MYC locus in genital tumours”. Oncogene. 25 (44): 5985–5993. doi:10.1038/sj.onc.1209625. PMID 16682952.
- ^ Mackay, Ian M.; Arden, Katherine E.; Nitsche, Andreas (15 tháng 3 năm 2002). “Real-time PCR in virology”. Nucleic Acids Research. 30 (6): 1292–1305. doi:10.1093/nar/30.6.1292. ISSN 0305-1048. PMC 101343. PMID 11884626.
Danh mục
[sửa | sửa mã nguồn]- Elyse; Houde, Alain (2002). “La PCR en temps réel: principes et applications” (PDF). Reviews in Biology and Biotechnology. 2 (2): 2–11. Bản gốc (PDF) lưu trữ ngày 12 tháng 6 năm 2009.
- Bustin, SA (2000). “Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays”. J Mol Endocrinol. 25 (2): 169–193. doi:10.1677/jme.0.0250169. PMID 11013345.
- Higuchi, R.; Dollinger, G.; Walsh, P.S.; Griffith, R. (1992). “Simultaneous amplification and detection of specific DNA-sequences”. Bio-Technology. 10 (4): 413–417. doi:10.1038/nbt0492-413. PMID 1368485. S2CID 1684150.
- Holland, P.M.; Abramson, R.D.; Watson, R.; Gelfand, D.H. (1991). “Detection of specific polymerase chain reaction product by utilizing the 50 !30 exonuclease activity of Thermus aquaticus DNA polymerase”. Proc. Natl. Acad. Sci. USA. 88 (16): 7276–7280. Bibcode:1991PNAS...88.7276H. doi:10.1073/pnas.88.16.7276. JSTOR 2357665. PMC 52277. PMID 1871133.
- Kubista, M; Andrade, JM; Bengtsson, M; Forootan, A; Jonak, J; Lind, K; Sindelka, R; Sjoback, R; Sjogreen, B; Strombom, L; Stahlberg, A; Zoric, N (2006). “The real-time polymerase chain reaction”. Mol. Aspects Med. 27 (2–3): 95–125. doi:10.1016/j.mam.2005.12.007. PMID 16460794.
- Higuchi, R.; Fockler, C.; Dollinger, G.; Watson, R. (1993). “Kinetic PCR: Real time monitoring of DNA amplification reactions”. Biotechnology. 11 (9): 1026–1030. doi:10.1038/nbt0993-1026. PMID 7764001. S2CID 5714001.
- Filion, M. (2012). Quantitative Real-time PCR in Applied Microbiology. Caister Academic Press. ISBN 978-1-908230-01-0.
- Wawrik, B; Paul, JH; Tabita, FR (2002). “Real-time PCR quantification of rbcL (ribulose-1,5-bisphosphate carboxylase/oxygenase) mRNA in diatoms and pelagophytes”. Appl. Environ. Microbiol. 68 (8): 3771–3779. Bibcode:2002ApEnM..68.3771W. doi:10.1128/aem.68.8.3771-3779.2002. PMC 123995. PMID 12147471.
- Logan J; Edwards K; Saunders N biên tập (2009). Real-Time PCR: Current Technology and Applications. Caister Academic Press. ISBN 978-1-904455-39-4.
